美国宾夕法尼亚大学G. R. Bowman教授小组JCTC论文:使用AlphaFold加速蛋白隐藏口袋的发现
2023-08-11 ComputArt计算有乐趣 ComputArt计算有乐趣 发表于上海
本项工作提出了一种使用AlphaFold来加速蛋白隐藏口袋发现的方法。通过调整多序列比对信息,可以让AlphaFold为目标蛋白生成多样化的三维构象。
 Theory and Computation
Theory and Computation
隐藏口袋是没有配体作用时,蛋白结构中不存在或不明显的一类可供药物结合的位点。从药物开发角度来看,瞄准这些神秘口袋是极具吸引力的。一方面,它可以让许多因缺少可药口袋而无法开发的靶点重获开发可能;另一方面,隐藏口袋通常远离活性位点,具有开发变构调节剂或高选择性药物的潜力。然而,发现和表征隐藏口袋也面临着诸多挑战。它们通常需要蛋白发生罕见的构象变化才能暴露,所以在实验测定的环境下很难观察到。并且,即使在分子动力学模拟中花费大量的计算资源,也不一定能采样到这类稀有的结构变化。
为此,来自美国宾夕法尼亚大学的Gregory R. Bowman教授团队提出了一套基于AlphaFold的隐藏口袋发现方法。该方法通过调整输入的多序列比对信息(MSA),让AlphaFold能够为目标蛋白生成多样化的三维构象,从而增加对隐藏口袋的采样几率。而对于隐藏口袋尚未完全暴露的情况,则进一步以生成的构象作为起始构象进行分子动力学模拟,并通过马尔科夫状态模型分析结果。在10种已知含有隐藏口袋的蛋白上,研究者展示了如何利用这套方法来加速隐藏口袋发现和表征的过程。相关研究成果近期发表于美国化学会出版的理论化学与计算化学核心期刊Journal of Chemical Theory and Computation【1】。
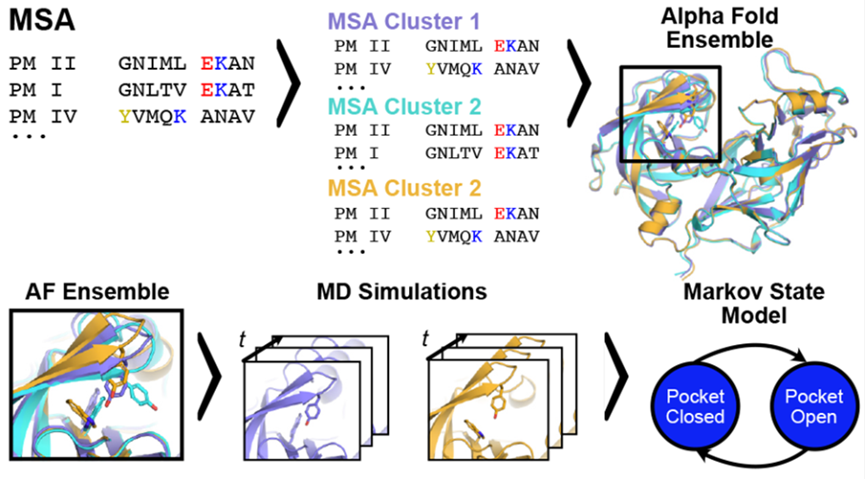
在方法实现的细节上(图-1),研究者首先根据查询序列搜集多序列比对信息(MSA),随后对MSA信息进行随机聚类并输入AlphaFold进行结构预测。MSA的作用是提供蛋白质序列的进化和保守信息,帮助AlphaFold生成更准确的三维结构。随机聚类则是为了让每次输入的MSA信息略有不同,从而使AlphaFold为查询序列生成多样的蛋白构象。在获得的构象集合中,可能已经包含了各种状态的隐藏口袋,通过将这些构象作为起始构象进行分子动力学模拟,最后再使用马尔可夫状态模型分析模拟结果,能够获得反映隐藏口袋各种状态的自由能形貌图,以帮助我们更好的发现与表征蛋白隐藏口袋。
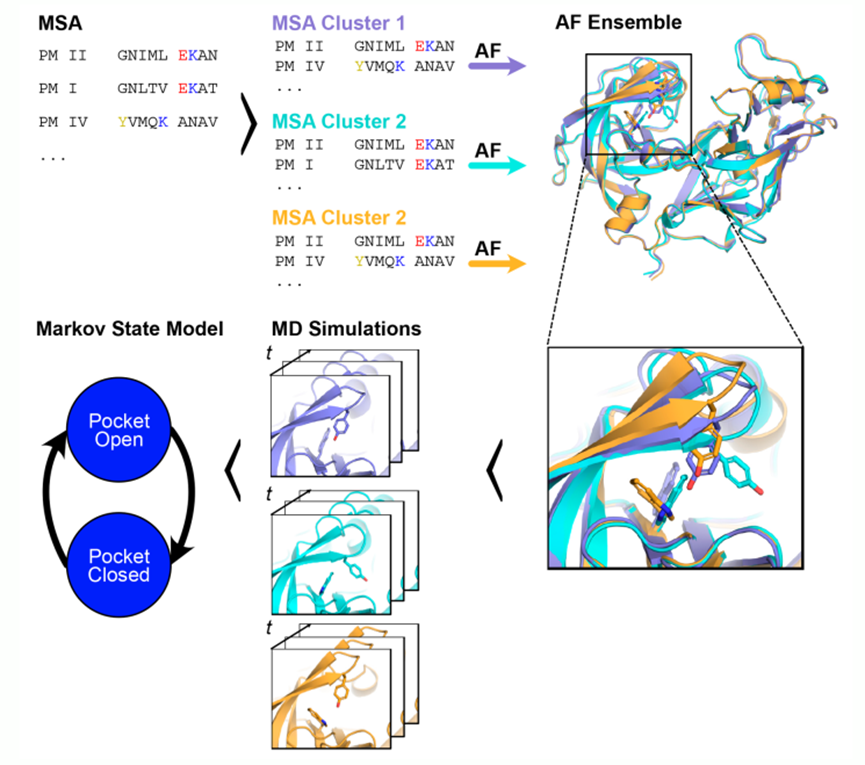
图1:基于AlphaFold的隐藏口袋探索流程(以PM II蛋白为例)
为考察该方法对蛋白隐藏口袋的采样能力,研究者整理了10个已知具有隐藏口袋的蛋白样本,这些蛋白需要经过一定的结构重排才能暴露隐藏位点。在评测过程中,首先使用AlphaFold为每个蛋白序列生成160个构象。然后,将预测结构与holo结构(隐藏口袋有配体结合的蛋白晶体结构)关于隐藏口袋所在区域的构象进行比较,若两者的RMSD小于1.2 Å,则认为AlphaFold为该样本成功预测出了开放状态的隐藏口袋。研究结果显示,Alphafold能够为10个样本中的6个样本成功采样出隐藏口袋呈开放状态的蛋白构象(图-2)。尤其是这10个样本中有5个holo结构未被Alphafold训练集所收录,生成构象能够重现其中的3种,充分说明了这套方法良好的采样与泛化能力。
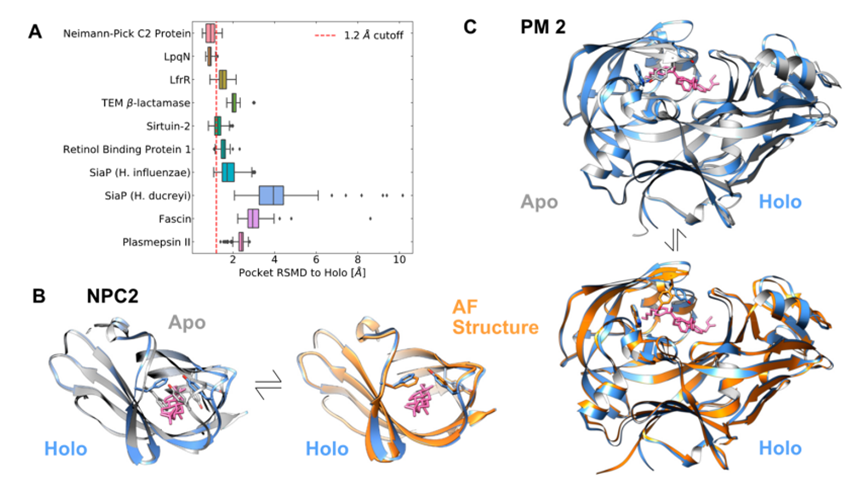
图 2: AlphaFold在10个样本上对隐藏口袋的采样表现
在上一步结果中,AlphaFold仅为PM2蛋白预测出了半开放式的隐藏口袋(图-2C)。研究者进一步将该蛋白所有的生成构象作为起始构象进行分子动力学模拟,并使用马尔科夫状态模型来分析模拟结果。同时,为了进行比较,以PM2蛋白的apo构象作为起点进行相同总时长的常规分子动力学模拟。已知PM2蛋白暴露隐藏口袋需要经过三种结构变化:Trp41改变侧链朝向,Tyr77沿χ-1翻转和“挡板”结构离开活性位点(图-3A)。结果显示,常规办法即使经过了32 微秒总时长的动力学模拟也无法采样出开放的PM II隐藏口袋,模拟过程中始终无法观察到 Trp41 侧链的翻转。而使用 AlphaFold 生成构象作为起点的动力学模拟则可以采样到全部的结构变化,能够成功观察到开放的隐藏口袋。
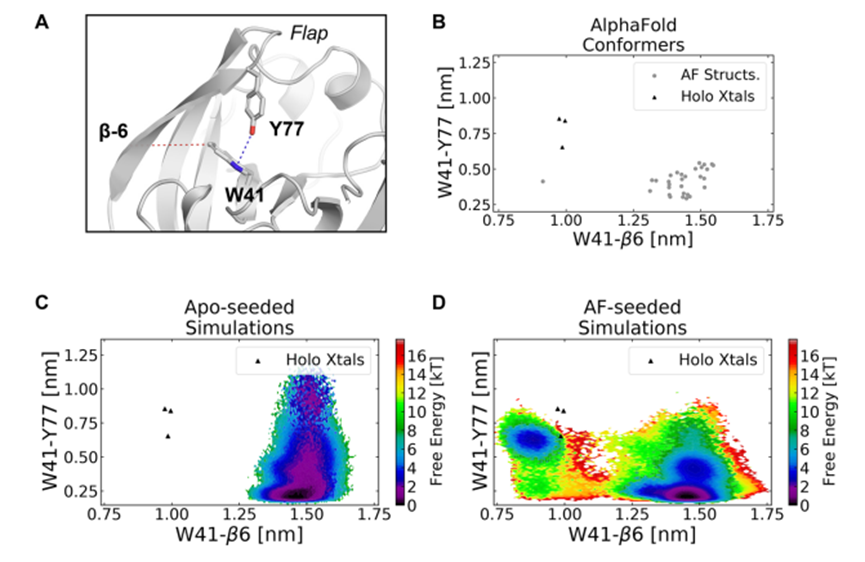
图3:以AlphaFold生成构象作为起点或apo构象作为起点的不同分子动力学模拟结果
【小结】本项工作提出了一种使用AlphaFold来加速蛋白隐藏口袋发现的方法。通过调整多序列比对信息,可以让AlphaFold为目标蛋白生成多样化的三维构象。在此基础上,进一步结合分子动力学模拟,可以相比常规方法更好的探索蛋白隐藏口袋。这种高效的隐藏口袋表征策略将有望为药物发现提供新的思路。
参考文献
【1】Meller A, Bhakat S, Solieva S, et al. Accelerating cryptic pocket discovery using AlphaFold[J]. Journal of Chemical Theory and Computation, 2023, 19, 14, 4355–4363
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言











