《自然》子刊:癌症基因诊断治疗新方法!
2023-08-20 小桔灯网 小桔灯网 发表于上海
作者开发了一种独特的DNA切除工具,该方法基于一种竞争性的DNA结合和消化机制,由单链磷硫酰化DNA引导的脱氧核糖核酸酶作用,高效去除野生型DNA链。
癌症的分子诊断强调了驱动基因改变在肿瘤生长和转移中的作用。检测和量化血液样本中cfDNA的基因组变化具有方便、微创和对早期肿瘤的高敏感性等优势。但cfDNA的突变通常非常低水平,这对现有的方法仍然是一个巨大的挑战。液滴数字PCR(ddPCR)可以定量检测丰度>0.1%的突变。下一代测序(NGS)具有较高的测序深度,可以检测到丰度低至10-7的突变。然而,这些方法的临床应用进一步受到探针设计复杂/温度稳健性要求,昂贵的辅助仪器/试剂以及易出现假阳性结果的限制。
检测低水平变异DNA一个主要挑战是来自大量WT DNA链的强背景。近日,一组来自中国的研究团队在杂志nature biomedical engineering上发表了一篇题为“Detection of low-frequency mutations in clinical samples by increasing mutation abundance via the excision of wild-type sequences”的文章。在这篇文章中,作者开发了一种独特的DNA切除工具,该方法基于一种竞争性的DNA结合和消化机制,由单链磷硫酰化DNA引导的脱氧核糖核酸酶 (sgDNase)作用,高效去除野生型DNA链。该方法在活检样本中发现了几个初始丰度为0.01%甚至更低的阳性突变,这些突变是NGS或全外显子测序(WES)无法检测到的。sgDNase具有单核苷酸分辨率和对所有序列的普遍适用性,可用于各种癌症和相关疾病的诊断和治疗。

图片来源:Nature biomedical engineering
单核苷酸分辨率sgDNase的构建
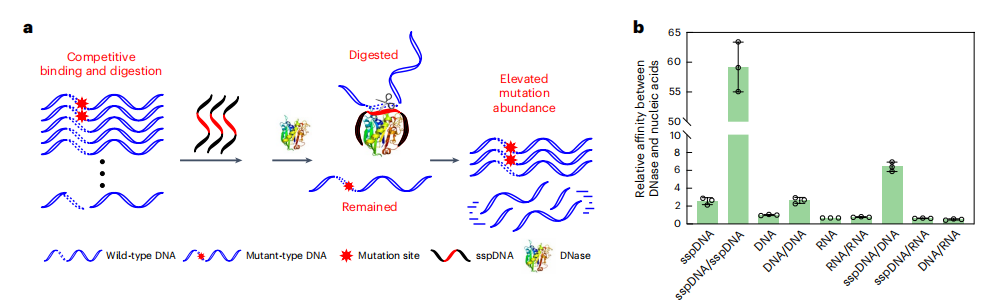
DNase与单链磷硫酰化DNA(sspDNA)预组装产生的复合物,仅会快速消化与sspDNA完全匹配的链。作者首先证实了sspDNA与DNase的结合亲和力高于正常DNA链(下图b)。
EGFRL858R突变是癌症最重要的驱动基因之一。采用S1-EGFR作为sspDNA链,FAM/ bhq1标记的EGFR L858R的WT链和MT链(P1-EGFR WT和P1-EGFR MT)分别作为完全匹配和单核苷酸错配的靶序列,结果证明了sgDNase的单核苷酸分辨能力,EGFR WT链几乎完全降解,而EGFR MT链基本保持完整。且随着变异等位基因频率(VAFs)从60%下降到0.3%,Discrimination factor增加了369倍,这意味着在非常低的VAFs样品中,sgDNase在单核苷酸分辨率下消除大量WT DNA链的性能更好。
sgDNase的概念证明。
图片来源:Nature biomedical engineering
通过sgDNase预处理富集热点区域的单个和多个突变
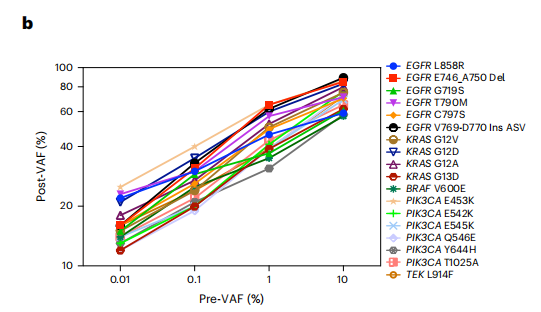
对于具有不同初始VAFs的合成样品中的EGFRL858R突变,经过sgDNase预处理后,VAFs分别从0.01%、0.1%、1%、10%和50%增加到22%、30%、46%、59%和95%。所有18个测试的不同靶突变,从pre-VAFs分别为0.01%、0.1%、1%和10%,而post-VAFs 分别为>10%、>18%、>30%和>50%,表明sgDNase体系对各种靶序列有着普遍适用性和强大的富集能力(如下图)。
在一些突变热点可包含数个不同的点突变,每一个点突变都可能发挥预测和/或预后作用。结果显示只要去除多余的WT链,所有不同类型的突变DNA链会同时富集。KRAS G12A、G12V、G12D混合物在pre-VAF为0.01% ~ 1%的范围内均富集到>10%的post-VAF,且sgDNase在较宽的温度范围内(34℃~ 45℃)均表现出较高的富集效率,进一步证明了sgDNase在温和温度条件下的适用性。
sgDNase预处理富集单一和多重热点突变。
图片来源:Nature biomedical engineering
多路sgDNase预处理
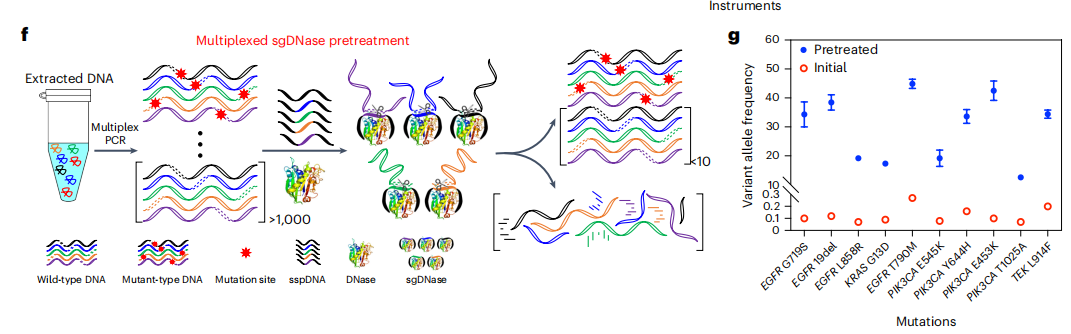
作者尝试使用多个sspDNA链同时富集临床相关靶突变的不同组合,进行多重sgDNase预处理(下图f)。作者使用EGFR、KRAS、PIK3CA和TEK基因中10条sspDNA链TBR内所有可能突变的多重富集。经过2小时的sgDNase预处理后,10个靶突变(EGFRL858R、EGFR19del、EGFR G719S、EGFRT79OM、KRASG13D,PIK3CAE545K,Y644H,E453K,T1025A和TEKL914F)都从低于0.3%(0.07%-0.27%)的pre-VAFs富集到高于10%的post-VAFs(12.4%到43.1%)。
多重sgDNase预处理的性能。
图片来源:Nature biomedical engineering
双sspDNA引导DNase预处理gDNA
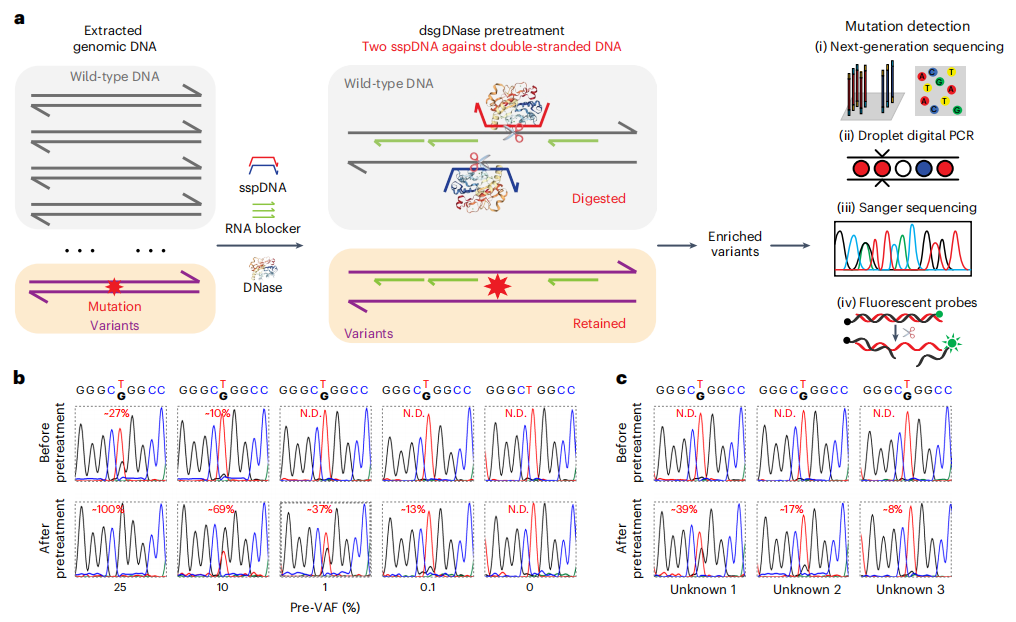
研究团队应用sgDNase直接预处理人gDNA(dsgDNase)。如下图a所示,从临床样品中提取的gDNA被剪切成DNA片段,并与一对目标sspDNA链混合。在加热和快速退火后,加入DNase快速消化完全匹配的WT dsDNA。经过dsgDNase预处理后,富集的变异可以很容易地通过各种方法检测,例如(1) NGS,(2) ddPCR法,(3)Sanger测序法,(4)荧光法。
结果显示在3个未知gDNA样本检测EGFRL858R突变,未经dsgDNase预处理时,Sanger测序结果中未观察到突变(下图c)。预处理后,突变为Sanger测序明确检测到,post-VAFs分别为39%、17%和8%。对于pre- VAFs低于0.01%的临床相关gDNA样品,使用gDNA的PCR扩增产物进行dsgDNase预处理后,样品的VAF丰度从0.01%显著富集至0.8-5.0%,易于用NGS检测。以上结果均证明了dsgDNase在gDNA直接预处理中对超低水平突变的敏感检测的可靠性。
dsgDNase对gDNA的直接预处理提高低水平突变的检出。
图片来源:Nature biomedical engineering
应用sgDNase预处理检测临床生物系统样品中的超低丰度突变
研究团队将sgDNase检测应用于70多个临床样本,共鉴定出16个阳性样本(见下图)。
未经sgDNase预处理时,只有2个组织样本被观察到EGFRL858R突变阳性,而没有检测到阳性血液样本。经过sgDNase预处理,3个组织样本和2个血液样本被鉴定为L858R突变阳性(下图a)。同样,图c、d比较了sgDNase预处理前后KRAS G12/G13突变的结果。结果显示经sgDNase预处理后,均检测出了之前未检测到的KRAS突变。将该方法扩展到检测静脉畸形患者活检标本中的突变(图e,f)。经过sgDNase预处理,作者在7个患者中检测出了PIK3CA的多种突变及TEKL914F突变。原始突变水平估计为1%或更低,与WES结果一致。
总的来说,在总共231个基因座中(肺癌患者17个样本,每个样本4个位点;结直肠癌患者38个样本,每个样本4个位点;静脉畸形患者11个样本,每个样本1位点,图g,h),观察到94.74% (18/19)的临床敏感性和98.58% (209/212)的临床特异性。
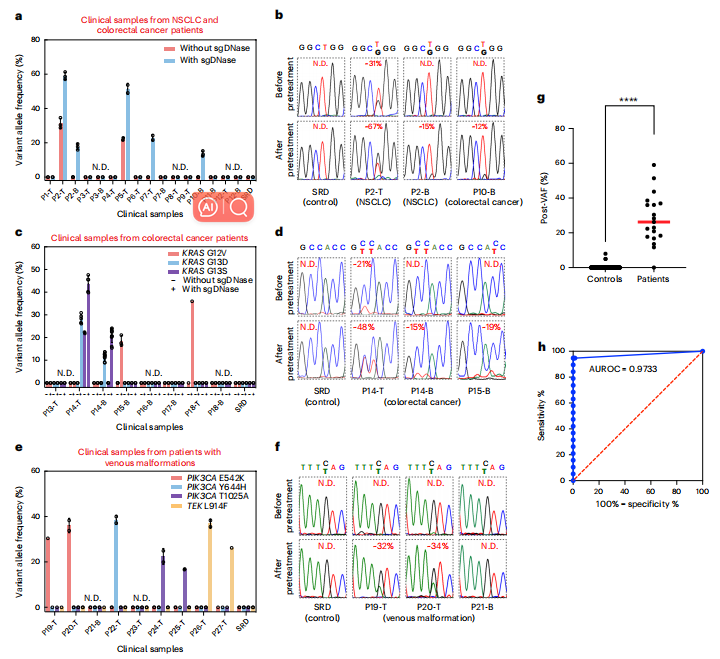
sgDNase预处理的临床生物系统样品中超低丰度突变检测。
图片来源:Nature biomedical engineering
总结与讨论
作者报告了一种强大的DNA切除方法sgDNase,用于在单核苷酸分辨率下检测基因组DNA和循环无细胞DNA中的低频突变。该方法采用了竞争结合-消化机制来引导sspDNA对DNase的序列特异性,显著提高了对单核苷酸变异的辨别能力。sgDNase预处理可富集初始频率低至0.01%的突变链,并对多序列背景下所有类型的单核苷酸错配有高辨别。该方法可与下一代测序、液滴数字聚合酶链反应、Sanger测序、荧光探针检测等多种突变检测方法相结合。
与其他富集方法相比,sgDNase具有以下优点:(1)预处理过程中sspDNA的量保持不变,允许突变链以不同的初始丰度进行充分富集。(2)sgDNase对不同的靶突变具有普遍适用性,没有任何序列限制。sspDNA的序列可以很容易地设计而不需要额外的修改。(3) sgDNase在温和的温度条件下工作良好,不需要严格的温度控制,也不需要长时间的高温培养。该方法成本低,易于实现,有利于保持DNA序列的原始信息。
本网站所有内容来源注明为“梅斯医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“梅斯医学”。其它来源的文章系转载文章,或“梅斯号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言







